小麦根系关系到养分吸收、转运、植株固定及代谢物合成、存储等生长功能[1]。根系表型分析有助于理解小麦在不同生长条件下的根型调控机制[2]。同时,小麦根系表型受遗传与环境双重调控,展现出个体多样性和环境适应性[3]。因此,研究小麦根系表型对于揭示其生长机理及优化栽培策略具有重要意义。
然而,根系主要生长于地下,这为根系研究带来挑战。20世纪后半叶,分根移位法和同位素标记法等方法得以应用,但这些方法存在损伤根系或精度不足的问题[4]。21世纪,光学相机与机器视觉技术的发展为根系研究提供了新的途径。二维图像分析根系表型时易受土壤遮挡影响[5-10],难以准确观测根系构型,限制了表型分析的精度[11]。因此,为提升分析准确性,构建三维根系模型正成为研究的新趋势[12]。
当前根系三维重建方法主要分为:三维形态模拟、三维扫描成像及计算机视觉三维重建3类。
三维形态模拟可以分析根系的拓扑结构与几何特征,建立动态模型来模拟其三维结构。其中基于L系统的根系几何结构模拟研究较为普遍,如小麦[13]、水稻[14]、水生植物[15]等。然而,此类三维模拟建模过程复杂,易受人为因素影响,且通常忽略个体差异,模拟精度有限。
三维扫描成像通常借助激光扫描技术、计算机断层扫描(Computed tomography,CT)或核磁共振成像(Nuclear magnetic resonance imaging,MRI)等成像方法,通过三维扫描重建根系模型[16-18]。该方法精度较高,但设备成本较高,重建效率较低,限制了其在高通量根系表型分析上的应用。
计算机视觉三维重建是基于多视角二维图像实现三维模型重建,重建方法涵盖轮廓投影、体素着色、运动恢复结构(Structure from motion,SFM)等。SFM算法由于具有使用简便、重建高效、算力要求较低的优势,近年来在农作物研究领域得到广泛应用,现有研究表明计算机视觉三维重建效率和精度较高,成本较低[19-21]。
本研究针对小麦根系高效三维重建需求,利用SFM算法对小麦根系进行三维重建并研究根系表型。
针对小麦根系3D表型高效提取需求,设计了一套专用小麦水培装置、换液装置及图像采集装置,并提出了相应的图像预处理方法,包括语义分割、畸变校正、折射校正、增设标志物,以提高重建精度。运用SFM-MVS算法构建小麦根系三维点云模型,并对点云进行稠密化,最后通过点云处理,提取根深、最大根长、根表面积和根体积等三维根系表型特征。研究总体技术路线图如图1所示。

图1 总体技术路线图
Fig.1 Overall technology flowchart
使用中国科学院遗传与发育生物学研究所提供的7种氮利用效率低(氮低效品种组)和7种氮利用效率高(氮高效品种组)共计14种不同小麦品种,通过催芽处理,4 d后所有种子萌发。将萌发的小麦种子移栽至水培装置中,在07:00—18:00打开全光谱生长补光灯,为小麦提供光照。整个生长过程在温室中进行,温室温度维持在15~18℃。为进一步探究不同氮水平的小麦根系生长差异,将每个品种的试验样本分为高氮组和低氮组,每组设立3个生物学重复,在移栽后第6天,配置相应的Yoshida高氮或低氮配方[22]作为小麦生长介质。
小麦水培装置结构如图2所示。本研究设计了一个直径20 cm、高40 cm、壁厚0.5 cm的圆柱型玻璃容器作为水培装置,并在其中注入水培营养液。此外,为确保小麦在不受损的情况下稳固生长,设计了一块厚1 cm、直径3 cm的圆柱型黑色海绵,并在海绵上切割一条狭缝来夹持小麦的茎基部。接着使用一块边长15 cm、厚度0.5 cm的正方形黑色牛皮纸覆面发泡板(Kraft paper laminated foam board,KT板),板中央剪出一个直径约2 cm的圆孔。将KT板的孔与海绵紧密配合,再将KT板固定于玻璃容器上,从而实现了小麦在水培装置中的稳固放置。
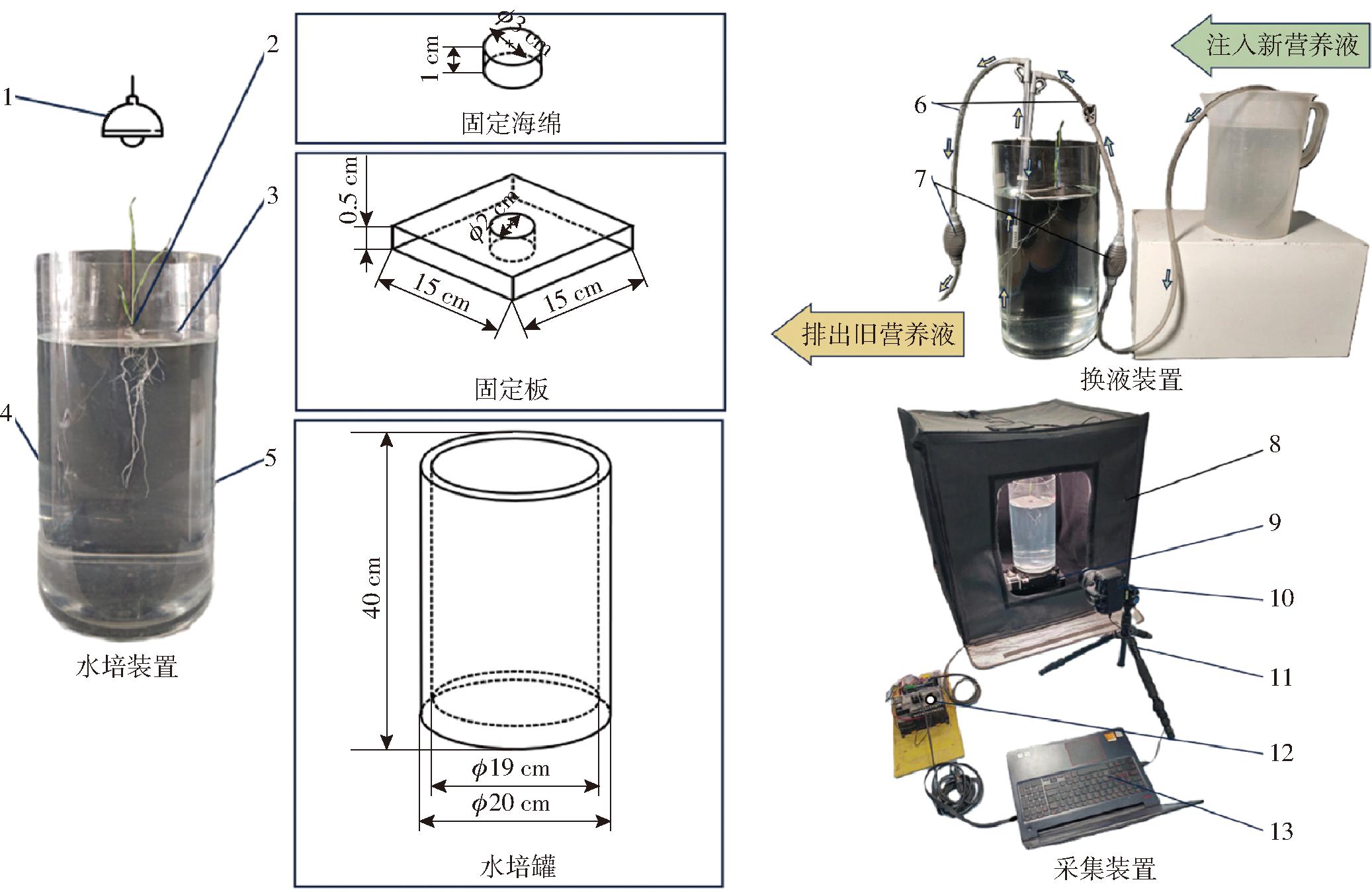
图2 水培、换液、采集装置结构示意图
Fig.2 Structural schematic of hydroponic,liquid exchange and collection devices
1.生长灯 2.固定海绵 3.固定板 4.营养液 5.水培罐 6.虹吸管 7.球型气囊 8.摄影棚、灯管 9.旋转台 10.相机 11.三脚架 12.PLC控制器 13.计算机
设计的配套换液装置结构如图2所示。小麦根部需要及时吸收养分,同时营养液久置会生长绿藻,不利于小麦培养及根系成像。同时,更换营养液可能导致根系结构产生巨大变化。因此,设计了一种柔和的换液方法及装置。该装置利用虹吸原理,通过配置于水培装置底部的虹吸泵排出旧营养液,同时,另一虹吸泵从备用容器中吸取新营养液并注入水培装置顶端。两个虹吸泵协同运作,流量相同,以维持水培装置内液位稳定,进而有效保持小麦根系的结构形态不发生大幅变化。
基于上述水培装置,设计了一套根系多视角图像采集装置,其结构如图2所示。该装置包括小型摄影棚、相机、三脚架、可编程逻辑控制器(Programmable logic controller,PLC)、电控旋转台和计算机。摄影棚顶部设置灯条作为光源;电控旋转台安装于小型摄影棚内部;水培装置放置于电控旋转台上;在摄影棚侧面的拍摄窗口处固定三脚架并安装相机,调节高度、俯角等拍摄位姿信息;最后调整并固定相机焦距,使用PLC控制旋转台带动水培装置一起旋转,同时相机从不同角度连续采集水培装置中根系的侧面图像。
从移栽第3天开始,使用根系图像采集系统,依据移栽后时间(Day after transplant,DAT)对所有试验植株的根系进行定期拍摄。在移栽7 d内,由于小麦根系发育迅速,每2 d采集1次;7~14 d根系生长速率减缓,每3 d采集1次;移栽14 d后,根系生长进一步放缓,每5 d采集1次。在小麦发育至分蘖期后,根系生长变化较小,因此于38 d后结束试验。为获取根系图像并提高采集效率,本研究采取视频抽帧技术,经试验,在确保模型精度与重建效率平衡的基础上,从每株小麦视频中选取150帧图像,用于后续的根系三维重建。
1.3.1 根系图像分割
Segformer神经网络基于Tranformer的优越性,展现出了优良的语义分割能力[23]。本文应用Segformer对根系图像进行分割,便于后续根系表型特征的提取。首先选取123幅视角不同,根系密度、深度和生物量情况差异大的根系图像作为训练集。使用Photoshop软件对训练集的图像进行像素级标注。将上述图像和对应的人工标注二值图输入Segformer-B2语义分割网络进行训练。学习率设为0.000 2,批次大小设为1,取训练迭代16 000次的模型作为根系语义分割模型。使用根系语义分割模型分割采集到的根系图像,得到根系二值图。基于语义分割结果二值图对根系图像掩膜处理,将图像中与根系无关的部分设为黑色,获得仅包含根系的图像。
1.3.2 图像畸变校正与折射校正
相机透镜固有的几何形态及其与相机传感器间的非平行配置,会导致采集的根系图像受到一定程度的径向与切向畸变影响,进而对后续根系分析造成干扰[24]。为应对此问题,本研究使用张正友标定法[25],将原始根系图像中的像素点逐一映射至校正后的坐标位置,从而对根系图像进行畸变校正。
畸变校正后,还需考虑光线折射导致的图像变形。在光线穿越水培装置的过程中,由于水和玻璃介质的介入,光线路径会产生折射现象,这会导致根系图像与真实情况之间存在显著的偏差,进而影响根系的3D重建。鉴于此,提出了一种针对该装置的折射校正技术,实现对根系图像的真实还原。
折射校正模型示意图如图3所示。图中,L1为相机到玻璃容器外壁的距离,t为玻璃容器厚度,L2为玻璃容器内壁到小麦根系中心距离,L为相机到小麦根系中心距离。α、β、γ分别为光线在空气、玻璃和水中传播时与水平面的夹角。点O为小麦根系三维空间中的真实点,Y为点O与相机光心的垂直距离,点O′为点O经光线折射后成像还原所在的三维空间位置点,点O与点O′之间的垂直距离为Δy,即折射造成的误差。L1、t、L2、L、Y、Δy单位均为mm。
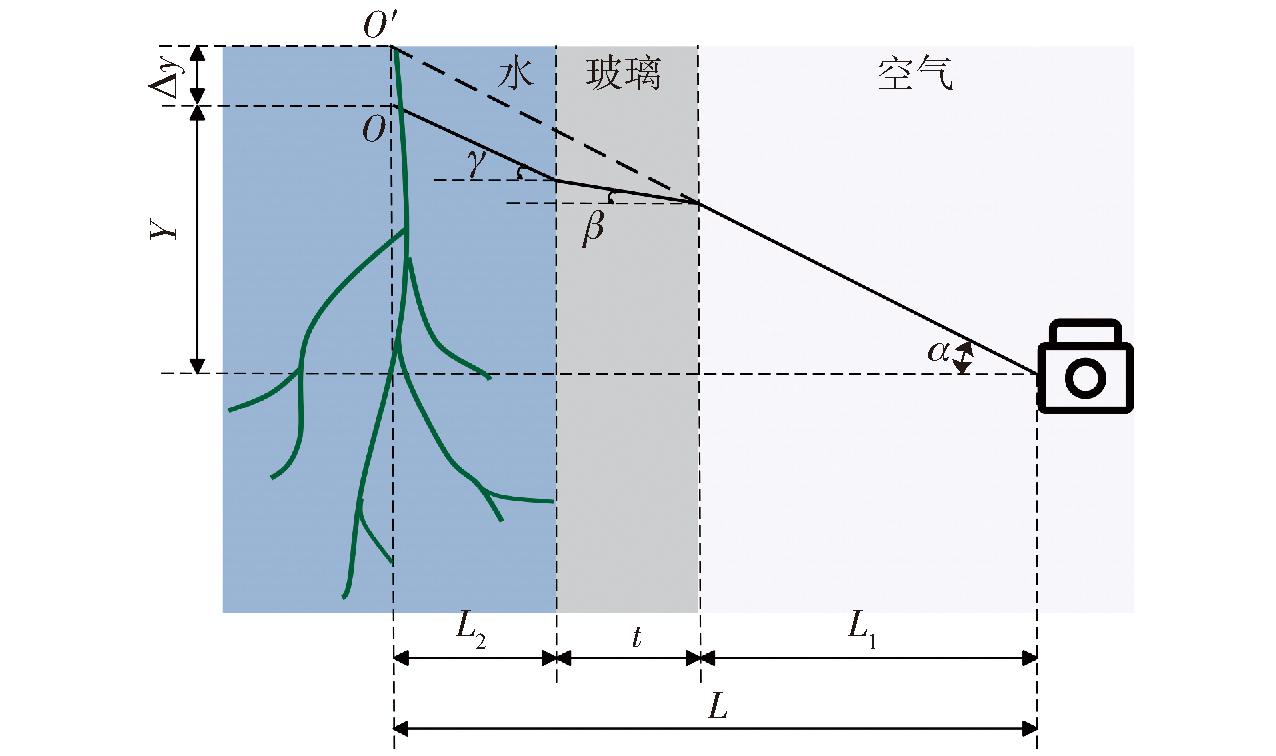
图3 折射校正模型示意图
Fig.3 Schematic of refractive correction model
考虑折射校正时,根系中心进入传感器的光线,视为垂直射入水面和玻璃面,未发生折射。与根系中心距离Y越大的点,在成像时光线进入玻璃和空气的入射角越大,发生折射偏移的距离Δy就越大。设空气、玻璃、营养液折射率分别为nα、nβ和nγ。本研究中,不考虑空气对光线的折射,不考虑营养物质对水折射率的影响,即将nα取值为1,nβ取值为1.5,nγ取值为水折射率1.33。考虑光线从玻璃进入空气时的折射和光线从营养液进入玻璃时的折射,根据斯涅尔定律,可得到折射角α、β、γ之间的关系式为
nαsinα=nβsinβ
(1)
nβsinβ=nγsinγ
(2)
根据勾股定理和正弦函数,可基于α、β、γ得到Y、Y+Δy与L的关系,并结合式(1)、(2)换算,可得
(3)
(4)
(5)
根据正切函数以及定义,可得L1、t、L2与L、Y关系式为
L=L1+t+L2
(6)
Y=L1tanα+ttanβ+L2tanγ
(7)
结合式(3)~(7),经换算,通过上述物理量求得折射误差Δy,计算式为
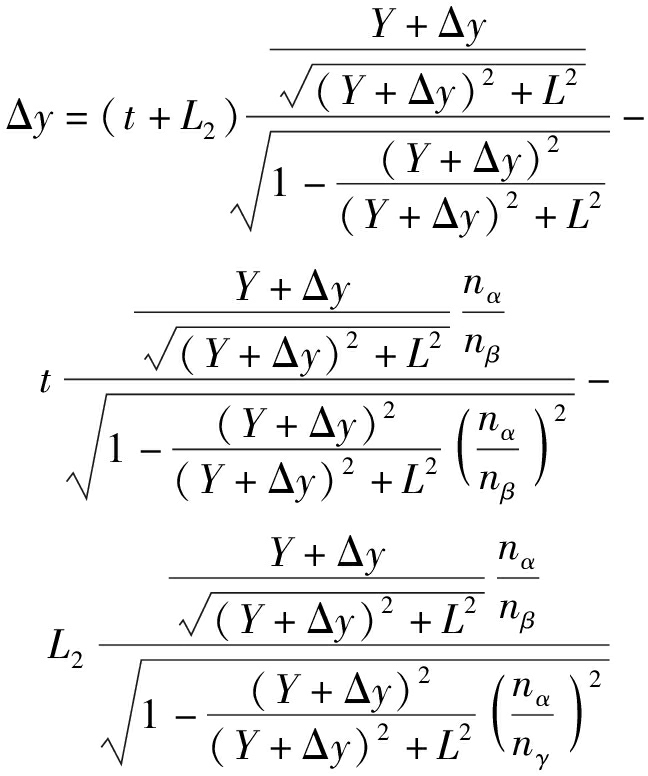
(8)
在试验时,考虑到Δy远小于Y,可认为(Y+Δy)2≈Y2,对式(8)进行简化,可得

(9)
为确定试验系统的空间分辨率,需建立像素尺度与实际物理长度的对应关系。根据相机成像参数,空间分辨率可通过每英寸像素数(Dots per inch,DPI)与单位换算关系计算得出。本试验相机DPI为72,可得到单像素物理尺寸计算式
(10)
式中,D为1个像素对应实际物理长度,单位为mm。经计算,根系图像中每个像素对应的现实世界长度为0.35 mm。
基于公式(9)、(10)可计算得到该像素点的折射偏差Δy。将每个像素点纵坐标减去Δy,以校正图像中像素点纵坐标位置。像素点横坐标校正过程与纵坐标校正过程相同。通过上述处理,获得折射校正后根系图像。
1.3.3 添加标志物辅助重建
小麦根系的每条主根颜色、粗细、纹理相近,这一特性给SFM算法的特征提取与匹配步骤带来了困难。单纯引入非相关像素点非但不能改进特征点的匹配效果,反而会引入噪声干扰。因此本研究提出了一种添加标志物策略,减小特征点提取与匹配的难度,且避免引入额外的噪声。
具体试验时,本研究准备若干标志物,标志物形状及颜色无特定限制,但每个标志物需有独特鲜明的形状、颜色与纹理特征,将它们贴附于小麦水培容器的顶端外壁一圈,不对小麦根系的生长及观测造成遮挡,为位姿估计提供参考。为提高试验效率,本研究仅对一个水培罐贴附标志物,在对该样本采样后,通过图像分割和裁剪,获得不同角度下的仅含标志物的图像,再将该组图像一一对应地覆盖至不同角度采集的其它小麦根系图像上,获得包含标志物的根系图像,接着进行三维重建,该过程如图4所示。
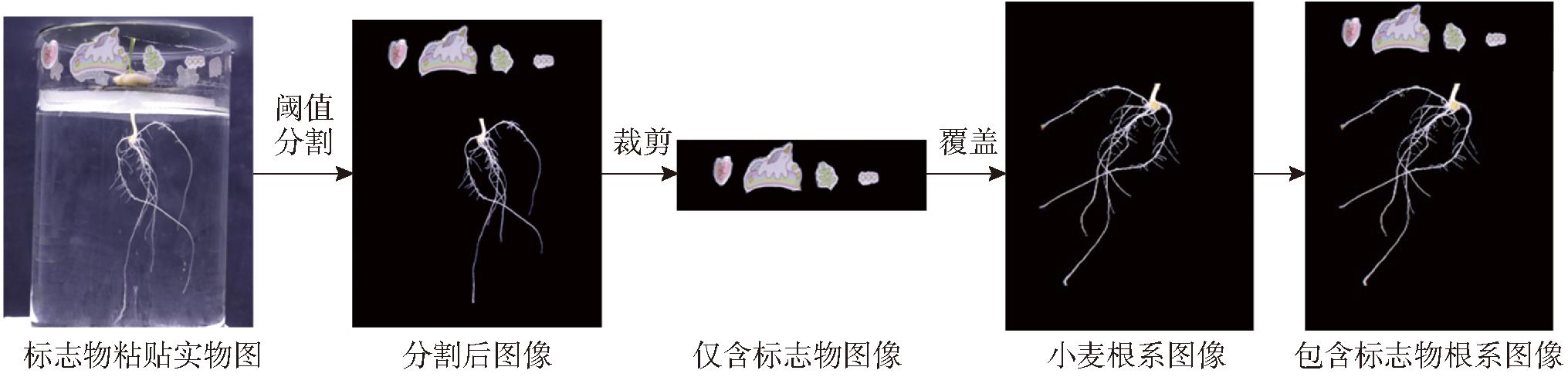
图4 标志物辅助重建流程图
Fig.4 Flowchart of landmark assisted reconstruction
SFM算法能够从图像序列中恢复出相机的运动轨迹及场景的三维结构。本研究采用SFM算法,基于多视角小麦根系图像,通过特征点提取与匹配,估计相机位姿,构建出小麦根系稀疏点云[26],接着通过光束平差法减小三维坐标点的误差[27-28],使用散列图像聚簇(Image clustering multi-view stereo,CMVS)与密集匹配(Patch match multi-view stereo,PMVS)方法[29]对点云进行稠密化,获得高精度的小麦根系三维模型。
对于三维重建得到的根系点云,本研究计算其顶端至底端的垂直距离来获得根深;将根系点云模型转化为面片集合和体素集合,分别计算所有面片面积总和与体素数量,获得根表面积和根体积。
设计了一种基于虹吸效应的换液装置,以减少换液对根系构型的影响。为评估基于虹吸效应的换液装置换液方法的有效性,将该方法与简单的换液方案(即将营养液全部排出再添加营养液的方法)进行对比。本试验通过对换液前后的根系进行图像采集,并对图像进行同样的分割处理以去除噪声,最后通过计算换液前后两幅图像之间的结构相似度指数(Structural similarity,SSIM)对根系构型相似度进行量化评估。SSIM值越接近1,说明两幅图像结构越相似。试验所使用图像及评估指标计算结果如图5所示。
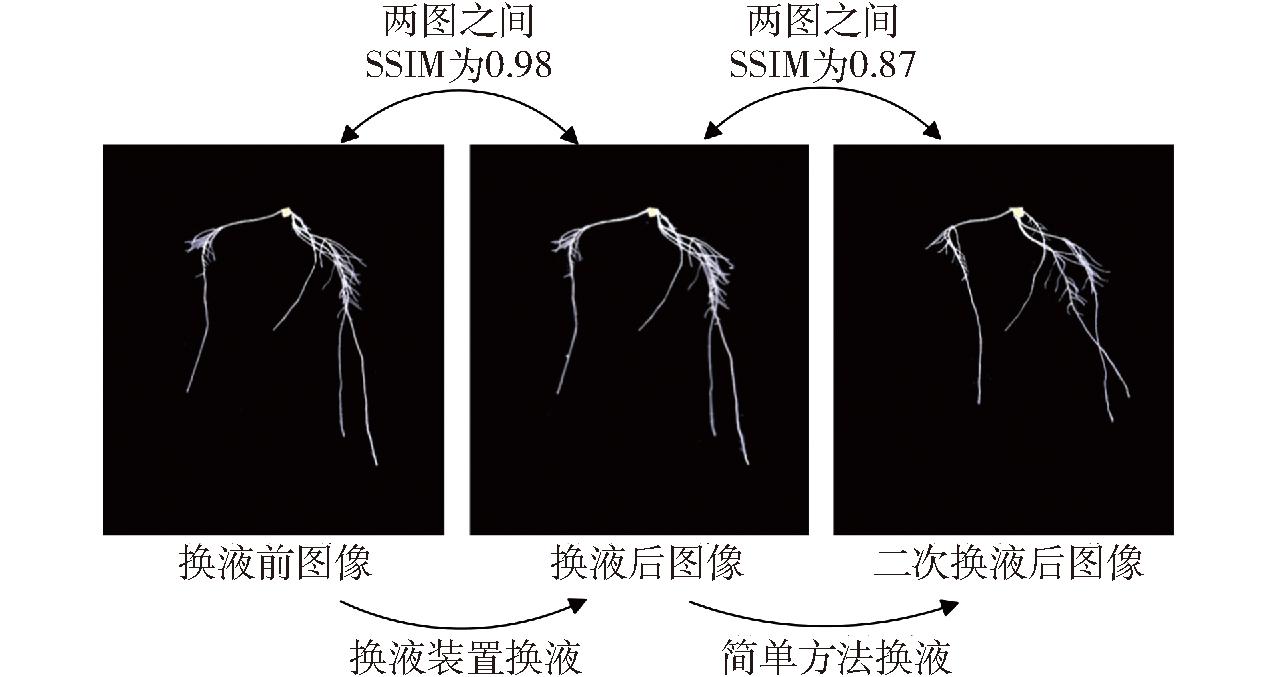
图5 换液前后根系构型对比
Fig.5 Comparison of wheat root system configuration before and after water change
由图5可知,使用本文提出的换液装置,换液前后的根型较为相似,这说明使用本研究提出的换液装置进行换液,由于换液更加舒缓,基本不会对根系构型造成影响,SSIM达到0.98。而简单换液方法先将水全部排出,根有一段时间直接裸露在空气中,由于失去了水的浮力,若干条主根会在重力作用下垂在一起,并容易相互黏附、缠绕,在重新加入营养液后,根在浮力作用下重新排布,构型因此会发生较大变化。通过观察简单方法换液前后两幅图,左侧的主根在换液后偏向右下方生长,右侧两条主根在换液后在下方出现了交叉,且SSIM值仅为0.87,说明简单的换液方法会对根系构型造成较大影响,进一步验证本研究提出的换液装置能够较好地维持根系构型。
2.2.1 折射校正准确性分析
为验证本研究提出的折射校正方法的准确性,设计了测试试验。将标定板置于水培玻璃容器内的水中,拍摄水中标定板图像IWater-Before,随后应用本研究的折射校正方法处理图像,获得校正后图像IWater-After。接着,将标定板移至相同高度和与相机相同距离的空气中拍摄,获得空气中标定板图像IAir。从标定板中随机选取6个内角点和6个标定格,测量IWater-Before、IWater-After、IAir 3幅图像中6个角点的像素坐标和6个标定格的对角线长度。结果显示,IWater-Before与IAir的像素点坐标平均相差25像素,而IWater-After与IAir的像素点坐标平均相差9.5像素,误差降低62%;IAir、IWater-Before和IWater-After平均对角线长度分别为61.1、71.3、64.7像素,校正后误差降低64.7%。IWater-Before与IAir像素的平均绝对误差为128.2像素,而IWater-After与IAir的平均绝对误差下降至115.9像素。基于上述3个评估指标可知,本研究提出的折射校正算法展现出良好的校正效果。
2.2.2 畸变校正和折射校正优化三维重建效果分析
为验证本研究使用的校正算法对三维点云重建精度的影响,设计了对比试验。对同一组试验数据进行不同的校正处理,并对不同处理的图像进行三维重建,统计点云数目,结果如图6所示。为进一步评估重建精度,计算重建点云中每个点到参考网格模型欧氏距离,并统计所有欧氏距离平均值(Mean Euclidean distance,MED)及欧氏距离标准差(Euclidean distance standard deviation,EDSTD)。平均值反映了重建点云与参考模型之间的整体偏差,其值越小,表明重建结果的整体精度越高;标准差则表征了点云与网格间距离的离散程度,其值越小,表示点云与网格的偏差越均匀,误差分布越一致,重建结果对细节的还原能力越强。

图6 不同校正对三维点云重建效果
Fig.6 Different calibration methods for 3D point cloud reconstruction
图6显示,基于未经校正的图像重建得到的点云数目最少,且由于成像偏差较大,根系出现明显的重影现象,如图中红框标注所示;而使用经过校正的图像重建的点云数目都会增多,根系重影问题有所缓解。这说明使用校正后的图像进行重建能显著提高小麦根系三维点云模型的精度。使用畸变校正对三维重建精度提升有限,点云左侧第2条主根有明显的折射造成的重影现象,而使用折射校正后,不会出现重影现象,且得到的点云数目更多,断根情况较少,对根系三维重建效果提升更明显。
使用5个3D打印的类根系模型,使用不同的校正方法及顺序进行重建,并通过ReeYee结构光三维扫描仪获得参考网格模型,接着计算重建点云的每个点到参考网格的欧氏距离平均值及标准差,来评估点云整体偏差以及离散程度。5个类根系模型评估结果平均值如表1所示。
表1 不同校正方法评估结果对比
Tab.1 Comparison of evaluation results of different calibration methods and sequences 像素
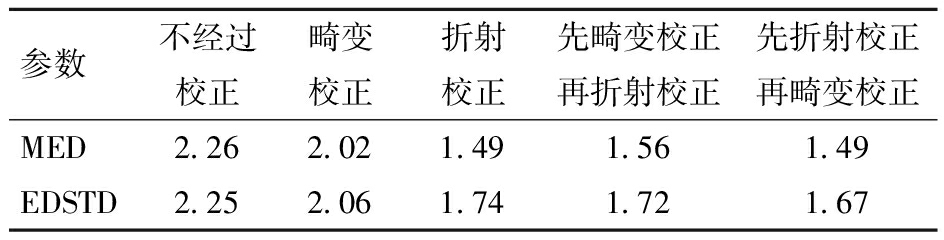
参数不经过校正畸变校正折射校正先畸变校正再折射校正先折射校正再畸变校正MED2.262.021.491.561.49EDSTD2.252.061.741.721.67
表1显示,总体上看,使用畸变校正后,相较于不使用校正方法,重建点云与参考网格的欧氏距离平均值和标准差都略有降低,说明使用畸变校正可以一定程度上改善根系点云的整体偏差以及离散程度。而使用折射校正方法后,两个评估指标显著降低,说明使用折射校正可以有效提高该重建方法的精度。另外,同时使用畸变校正与折射校正的重建精度与仅使用折射校正的重建精度较为接近,且畸变校正和折射校正的使用顺序对重建点云精度的影响较小。考虑到畸变校正和折射校正的作用不同,且点云数目一定程度上反映了点云精细度,点云数目越多,说明三维模型展示了更多细节,更加精细,因此本研究采用同时使用畸变校正和折射校正的方案。
上述结果说明在该采集与重建方案下,折射对点云精度带来的影响较大,本文提出的折射校正算法能够较好解决折射对图像的干扰,提升三维重建精度。对比其他结果而言,两种校正方法一起使用,重建出的点云主根几乎没有断裂现象,没有重影,模型最精细。这说明畸变与折射都影响了三维重建的精度,本文提出的畸变校正加折射校正的方案,能够有效获得精度较高的三维模型。
为提高三维重建精度,在多视角根系图像上添加了对应视角的标志物图像。为验证该方法的有效性,本文基于有无标志物的两组同一植株的图像,分别进行三维重建,并对比点云数目,结果如图7所示。
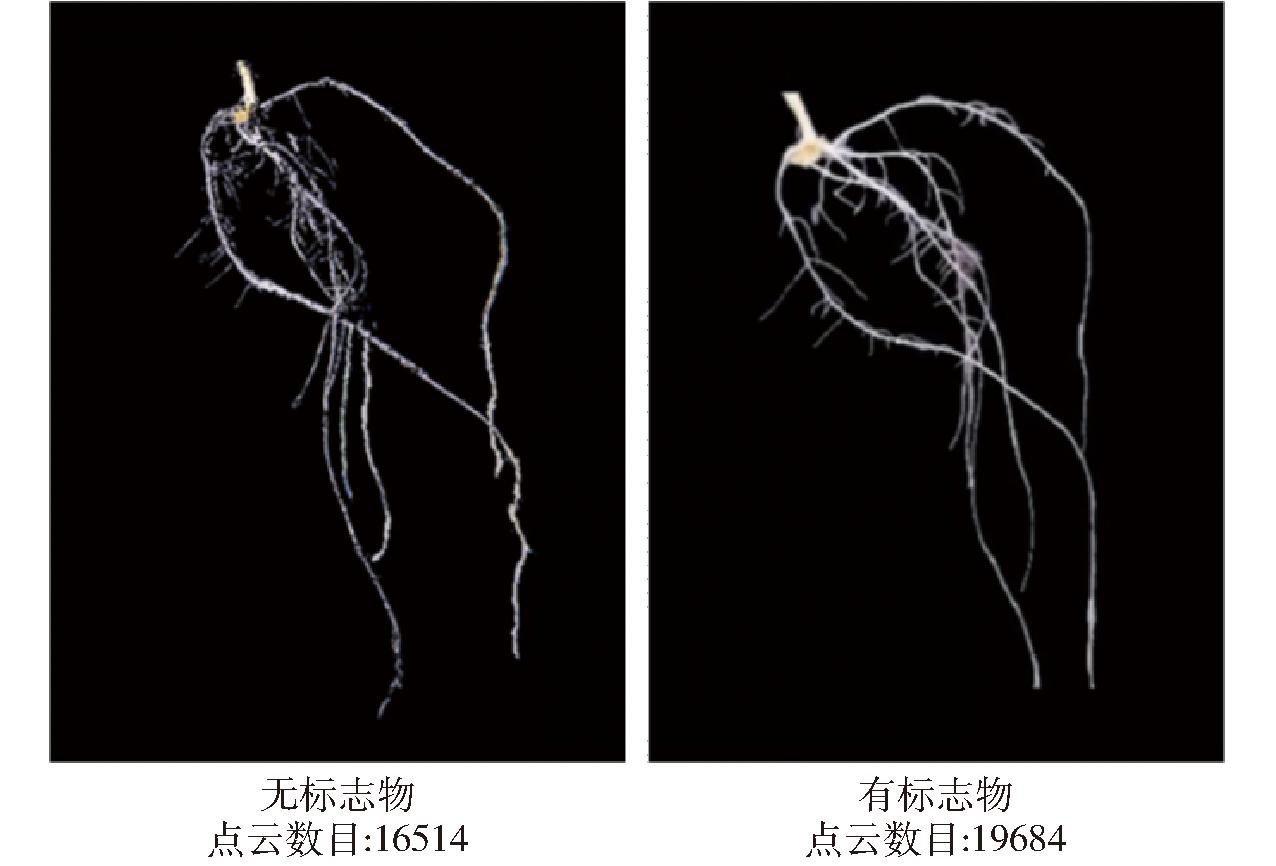
图7 有无标志物图像三维重建效果对比
Fig.7 Comparison of 3D reconstruction effects of images with and without biomarkers
结果显示,在不添加标志物的情况下,由于根系的多条主根表型特征非常相似,特征匹配较为困难,点云略显离散、模糊;而添加标志物后,由于标志物的特征明显,减小了匹配难度,重建出的三维点云呈现出的根系更加连贯、清晰。从量化指标上看,增加标志物的图像分辨率更高,重建耗时仅增加约3%,而点云数目增加约0.2倍。本文还使用2.2节所述的类根系模型进行评估,增加标志物的重建结果相较于不加标志物的结果,欧氏距离平均值从5.78像素降低至1.56像素,欧氏距离标准差从3.58像素降低至1.72像素,这说明重建点云的整体偏差显著减小,点云分布更加集中。因此,为多视角图像增加标志物可提高小麦根系三维模型的重建精度。
为进一步探究氮环境对小麦根系生长的影响,使用1.2节提出的采集方案对1.1节设置的低氮组和高氮组根系样本分别进行10次图像采集。图8展示了同一个品种的小麦在不同氮水平培养条件下的根系生长情况。

图8 不同氮水平小麦根系生长情况对比
Fig.8 Comparison of root growth of wheat at different nitrogen levels
图8显示,将小麦样本移栽至水培系统后前6 d内,两组试验对象在相同培养条件下,其根系发育情况未见显著差异。在施加了不同氮浓度营养液后,可以观察到低氮环境下小麦根系展现出更快的生长速率,其根系长度和深度增加,侧根也更加丰富。经过28 d的培养,低氮组根系几乎完全遍布培养容器,而在高氮组中,根系长度、深度以及侧根数量等表型特征显著低于低氮组。
为探究不同氮利用效率的小麦在高氮和低氮环境下根系的生长情况,本研究分别对7种氮低效品种和7种氮高效品种进行试验,将上述小麦样本分别在低氮和高氮环境下培养,并收集所有样本在10个不同时间点的三维表型数据,通过Z-score法去除每组生物学重复中的离群值,绘制了箱线图,并使用独立t检验对这些数据进行差异显著性分析,如图9所示,图中*表示0.01≤p<0.05,**表示0.001≤p<0.01,***表示0.000 1≤p<0.001,****表示p<0.000 1。
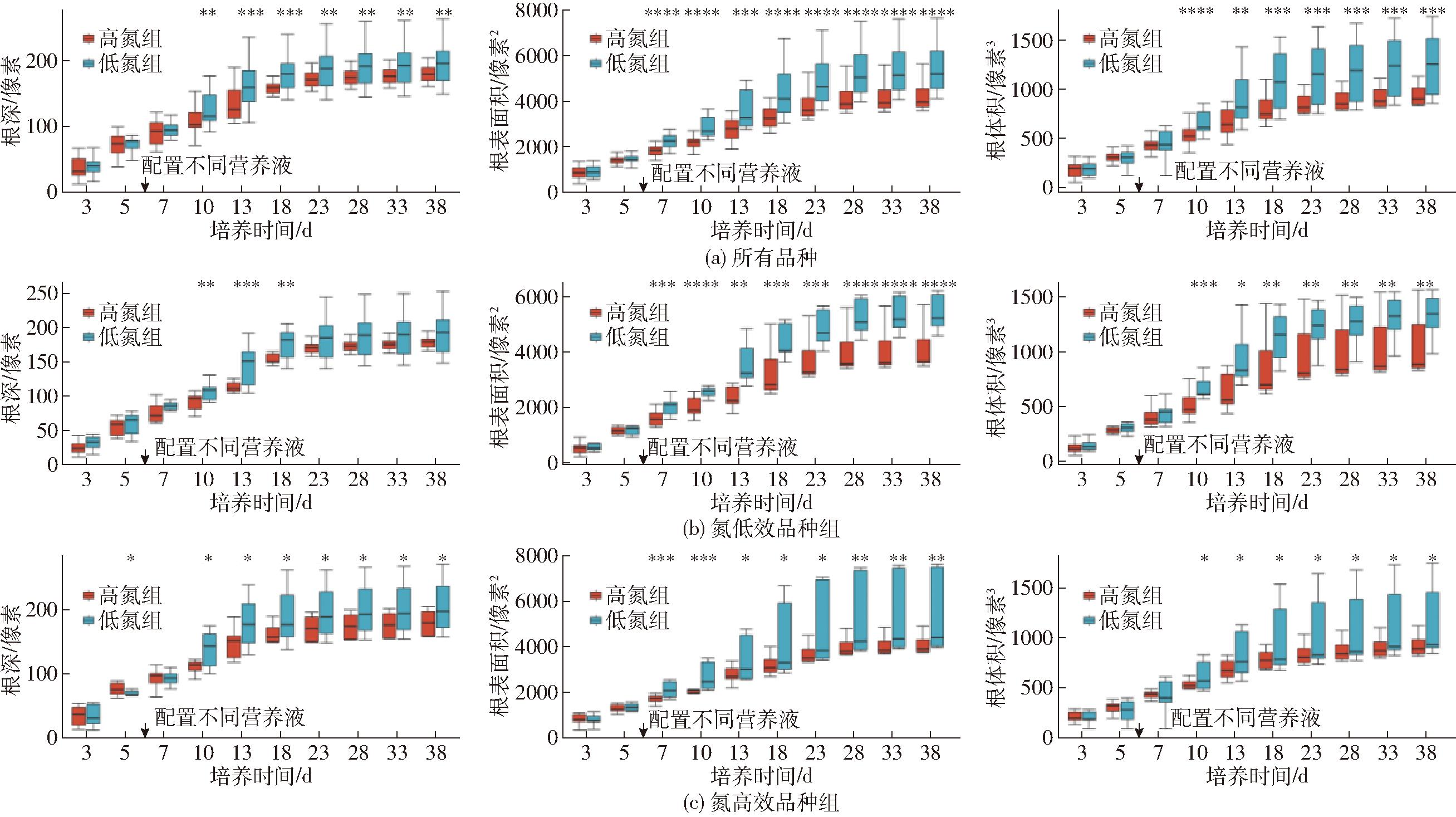
图9 不同氮环境下小麦根系表型箱线图
Fig.9 Box plots of root depth,root surface area and root volume of wheat roots at different nitrogen levels
结果显示,在配置不同营养液前,小麦生长前期,相同环境下植株之间根系的三维表型基本没有显著差异。在配置不同营养液后,随着时间推移,高氮组和低氮组在三维根系表型上都出现了显著差异,低氮组小麦根系的根深、根表面积和根体积普遍大于高氮组小麦根系,这说明在低氮环境下,小麦倾向于生长出更深、分布更密集的根系,以汲取足够的养分。
通过比较高氮组与低氮组在配置不同营养液后根系表型出现显著差异的时间,发现根表面积在第7天出现显著差异,根深和根体积均从第10天出现显著差异,这显示根表面积这一表型对氮环境的响应速度更快,小麦根系可能优先生长出较细的侧根,以快速适应环境变化,随后再发育出更深和粗壮的主根。
对比氮低效品种组和氮高效品种组,对氮环境变化的响应时间基本一致,但氮低效品种组的根深、表面积和体积在高氮和低氮下的差异更加显著,表现出更低的p值,这表明氮低效品种组对氮环境的变化更敏感。另外,在氮低效品种组中,根深这一指标从第23天后,高氮组和低氮组不再有显著差异。这表明氮低效品种可能在早期阶段更敏感,快速响应低氮环境,但在后期可能由于营养供应下降,根生长受到一定限制。而氮高效品种的根深在后期一直展示出显著性,可能是因为其对低氮具有更高的耐受性,在整个生长过程中都能维持较强的根系生长。
(1)针对传统小麦根系表型特征观测方法的局限性,开发了一套小麦水培装置和图像采集系统,实现根系生长观测,大幅提高了图像采集效率;设计了基于虹吸原理的换液方案及装置,维持根型稳定,有利于根系生长动态监测;针对装置对成像产生的折射和重建精度不足问题,提出了图像折射校正算法和标志物辅助重建方法,提高了三维重建精度;并设计了一套根系三维表型特征提取算法。
(2)改进的换液方法可使换液前后根系图像结构相似度提高至0.98;提出的折射校正算法对比未校正的根系图像,校正后图像像素点坐标与真实值差值降低62%,平均对角线长度与真实值差值降低64.7%,平均绝对误差从128.2像素下降至115.9像素;根系参考模型的评估试验表明,折射校正和增加标志物的方法均显著提高了根系重建点云的整体精度。
(3)利用研究的装置开展了小麦试验,结果表明低氮环境能促使小麦发育出更繁密的根系,表现出更深的根系和更大的根表面积及体积;相比于氮高效品种组,氮低效品种组对氮环境变化更敏感。
[1] SKAGGS T,SHOUSE P.Roots and root function:introduction[J].Vadose Zone Journal,2008,7(3):1008-1009.
[2] BEKKERING C,HUANG J,TIAN L.Image-based,organ-level plant phenotyping for wheat improvement[J].Agronomy,2020,10(9):1287.
[3] FRESCHET G,PAG S L,IVERSEN C,et al.A starting guide to root ecology:strengthening ecological concepts and standardising root classification,sampling,processing and trait measurements[J].New Phytol,2021,232(3):973-1122.
S L,IVERSEN C,et al.A starting guide to root ecology:strengthening ecological concepts and standardising root classification,sampling,processing and trait measurements[J].New Phytol,2021,232(3):973-1122.
[4] 程建峰,潘晓云,刘宜柏.作物根系研究法最新进展[J].江西农业学报,1999,11(4):55-59.CHENG Jianfeng,PAN Xiaoyun,LIU Yibai.The latest progress in crop root research methods[J].Acta Agriculturae Jiangxi,1999,11(4):55-59.(in Chinese)
[5] PATE A G,INGRAM K.Digital acquisition and measurement of peanut root minirhizotron images[J].Agronomy Journal,2000,92(3):541-544.
A G,INGRAM K.Digital acquisition and measurement of peanut root minirhizotron images[J].Agronomy Journal,2000,92(3):541-544.
[6] 吴长高,罗锡文.计算机视觉技术在根系形态和构型分析中的应用[J].农业机械学报,2000,31(3):63-66.WU Changgao,LUO Xiwen.Application of computer vision technology in root morphology and configuration analysis[J].Transactions of the Chinese Society for Agricultural Machinery,2000,31(3):63-66.(in Chinese)
[7] 刘九庆,汤晓华,陈健,等.基于线阵CCD植物微根系图像监测分析系统[J].林业科学,2005,41(3):121-124.LIU Jiuqing,TANG Xiaohua,CHEN Jian,et al.The plant fine roots image monitor analysis system based on linear CCD[J].Forestry Science,2005,41(3):121-124.(in Chinese)
[8] WANG M,ZHANG Q.Issues in using the WinRHIZO system to determine physical characteristics of plant fine roots[J].Acta Ecologica Sinica,2009,29(2):136-138.
[9] APTE A.Detecting piecewise linear networks using reversible jump markov chain monte carlo[D].Clemson:Clemson University,2010.
[10] WELLS D,FRENCH A,NAEEM A,et al.Recovering the dynamics of root growth and development using novel image acquisition and analysis methods[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2012,367:1517-1524.
[11] CHOUDHURY S,MATURU S,SAMAL A,et al.Leveraging image analysis to compute 3D plant phenotypes based on voxel-grid plant reconstruction[J].Frontiers in Plant Science,2020,11:521431.
[12] 史维杰,郝雅洁,赵明霞,等.基于三维重建技术的小麦表型分析[J].物联网技术,2019,9(9):84-86.SHI Weijie,HAO Yajie,ZHAO Mingxia,et al.Wheat phenotype analysis based on 3D reconstruction technology[J].Internet of Things Technology,2019,9(9):84-86.(in Chinese)
[13] 王美丽,何东健.基于L系统的小麦根系可视化模拟研究[J].农机化研究,2008,30(3):36-39.WANG Meili,HE Dongjian.Research on visual simulation of wheat root system based on L system[J].Journal of Agricultural Mechanization Research,2008,30(3):36-39.(in Chinese)
[14] 杨乐,彭军,杨红云,等.基于微分L系统的水稻根系三维生长模型研究[J].农业机械学报,2019,50(10):208-214.YANG Le,PENG Jun,YANG Hongyun,et al.Research on three dimensional growth model of rice root system based on differential L-system[J].Transactions of the Chinese Society for Agricultural Machinery,2019,50(10):208-214.(in Chinese)
[15] 黄晓龙,李松阳,李宽意.基于L系统的水生植物根系动态生长模型构建[J].湖泊科学,2022,34(6):2083-2094.HUANG Xiaolong,LI Songyang,LI Kuanyi.Construction of dynamic growth model of aquatic plant roots based on L system[J].Lake Science,2022,34(6):2083-2094.(in Chinese)
[16] FANG S,YAN X,LIAO H.3D reconstruction and dynamic modeling of root architecture in situ and its application to crop phosphorus research[J].The Plant Journal:for Cell and Molecular Biology,2009,606:1096-1108.
[17] MAIRHOFER S,ZAPPALA S,TRACY S,et al.RooTrak:automated recovery of three-dimensional plant root architecture in soil from x-ray microcomputed tomography images using visual tracking[J].Plant Physiology,2012,158(2):561-569.
[18] SCHULZ H,POSTMA J,DUSSCHOTEN D,et al.Plant root system analysis from MRI images[J].Communications in Computer and Information Science,2013,359:411-425.
[19] 柴宏红,邵科,于超,等.基于三维点云的甜菜根表型参数提取与根型判别[J].农业工程学报,2020,36(10):181-188.CHAI Honghong,SHAO Ke,YU Chao,et al.Extraction of sugar beet root phenotype parameters and root type discrimination based on three-dimensional point cloud[J].Transactions of the CSAE,2020,36(10):181-188.(in Chinese)
[20] 黎兆锴,宋亚男,徐荣华,等.基于SFM与深度学习融合的水果体积测量算法[J].计算机工程与设计,2023,44(6):1699-1705.LI Zhaokai,SONG Ya’nan,XU Ronghua,et al.Fruit volume measurement algorithm based on the fusion of SFM and deep learning[J].Computer Engineering and Design,2023,44(6):1699-1705.(in Chinese)
[21] 姚雨旸.基于旋转成像平台的作物根系三维重构方法[J].信息与电脑,2023,35(6):29-33.YAO Yuyang.3D reconstruction method of crop root system based on rotational imaging platform[J].Information and Computer,2023,35(6):29-33.(in Chinese)
[22] YOSHIDA S.Fundamentals of rice crop science[M].International Rice Research Institute:Los Ba os,Layuna,Philippines,1981.
os,Layuna,Philippines,1981.
[23] XIE E,WANG W,YU Z,et al.SegFormer:simple and efficient design for semantic segmentation with transformers[J].Curran Associates Inc,2021,924:12077-12090.
[24] ZELINSKY A.Learning OpenCV computer vision with the OpenCV library[J].IEEE Robotics &Automation Magazine,2009,16(3):100.
[25] ZHANG Z.A flexible new technique for camera calibration[J].IEEE Transactions on Pattern Analysis and Machine Intelligence,2000,22(11):1330-1334.
[26] SCHONBERGER J,FRAHM J.Structure-from-motion revisited[C]∥IEEE Conference on Computer Vision &Pattern Recognition,2016:4104-4113.
[27] 黄慧敏.基于多视角图像的三维重建技术研究[D].南京:南京师范大学,2020.HUANG Huimin.Research on 3D reconstruction technology based on multi perspective images[D].Nanjing:Nanjing Normal University,2020.(in Chinese)
[28] 黄身勇,蒋国平.基于多视角数字图像的深基坑三维重构研究[J].河南科技,2021,40(16):10-14.HUANG Shenyong,JIANG Guoping.Research on 3D reconstruction of deep foundation pit based on multi perspective digital images[J].Henan Science and Technology,2021,40(16):10-14.(in Chinese)
[29] FURUKAWA Y,PONCE J.Accurate,dense,and robust multiview stereopsis[J].IEEE Transactions on Pattern Analysis and Machine Intelligence,2010,32(8):1362-1376.